2. 教師なし学習¶
2.1. クラスタリング¶
K-means¶
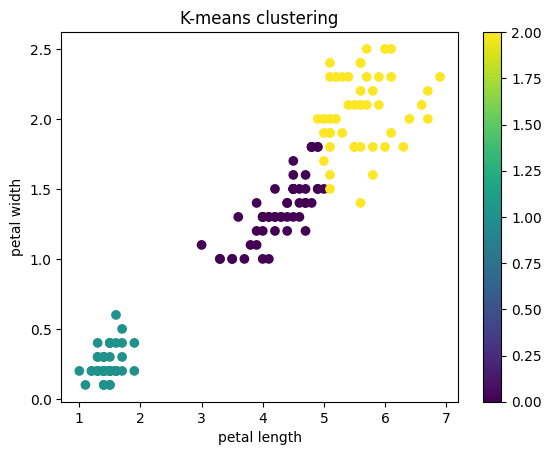
irisの特徴量petal_lengthとpetal_widthを用いて、クラスタリング手法の一つであるK-meansを使ってクラスタリングします
K-meansではクラスタの数を指定する必要があります。ここでは3つのクラスタを生成します。
[1]:
import matplotlib.pyplot as plt
import pandas as pd
from sklearn import datasets
from sklearn.cluster import KMeans
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
%matplotlib inline
[2]:
data = datasets.load_iris()
df = pd.DataFrame(data.data, columns=data.feature_names).reset_index(drop=True)
target = pd.DataFrame(data.target, columns = ['species']).reset_index(drop=True)
df = df.merge(target, left_index=True, right_index=True, )
df.head()
[2]:
| sepal length (cm) | sepal width (cm) | petal length (cm) | petal width (cm) | species | |
|---|---|---|---|---|---|
| 0 | 5.1 | 3.5 | 1.4 | 0.2 | 0 |
| 1 | 4.9 | 3.0 | 1.4 | 0.2 | 0 |
| 2 | 4.7 | 3.2 | 1.3 | 0.2 | 0 |
| 3 | 4.6 | 3.1 | 1.5 | 0.2 | 0 |
| 4 | 5.0 | 3.6 | 1.4 | 0.2 | 0 |
[3]:
X_iris=df[['petal length (cm)', 'petal width (cm)']]
model = KMeans(n_clusters=3) # k-meansモデル、n_clustersでクラスタの数を指定
model.fit(X_iris) # モデルをデータに適合
y_km=model.predict(X_iris) # クラスタを予測
[4]:
plt.figure()
plt.scatter(df['petal length (cm)'], df['petal width (cm)'], c = y_km)
plt.colorbar()
plt.xlabel('petal length')
plt.ylabel('petal width')
plt.title('K-means clustering')
plt.show()
PCA(主成分分析)次元削減¶
PCA(主成分分析)で次元削減を行う目的はいくつかあります。
主に以下のような理由です。
計算コストの削減
特徴量が多いと、機械学習モデルの学習に時間がかかります、過学習のリスクも高まります。
PCAで特徴量を減らすことで、学習時間の短縮やモデルのシンプル化が可能になります。
特徴量が減れば、学習・予測の速度が向上します。
情報の圧縮(次元削減)
データには多数の特徴量(変数)があることが多いですが、それらの中には似たような情報を持っているものもあります。
PCAは相関の高い変数をまとめ、「できるだけ情報を失わないように」少数の新しい軸(主成分)に圧縮します
→ 少ない次元で効率的にデータを表現できる。
→ 重要な特徴を抽出できる。
→ 予測に本当に役立つ要素だけを取り出すことができます。
データの可視化が容易になる
圧縮した2次元・3次元データなら、散布図などで傾向を直感的に把握できます。
PCAを使えば、高次元のデータを2次元や3次元に射影できるので、クラスタリングの傾向や異常値の存在などが見やすくなります。
ノイズ除去
高次元データの中には、分析にほとんど寄与しない「ノイズ的な変数」も含まれています。
PCAでは分散が小さい成分(=ほとんど情報を持たない軸)を落とすことで、ノイズを減らし、より本質的な構造を捉えることができます。
ここでは、irisデータの4つの特徴量を2次元にPCAを用いて削減します。
[5]:
X = df.iloc[:, :4]
y = df['species']
PCAを実行する前に標準化します。
[6]:
# 特徴量の標準化
X = StandardScaler().fit_transform(X)
[7]:
# PCAの実行(2次元に圧縮)
pca = PCA(n_components=2)
X_pca = pca.fit_transform(X)
[9]:
print(X.shape) # もともとの特徴量
(150, 4)
[10]:
print(X_pca.shape) # 特徴量が圧縮されている。次元が削減されている
(150, 2)
どのくらいデータを説明できているか、寄与率(explained_variance_ratio)を用いて確認します。
PCAの説明分散(または固有値)は、各主成分に帰属させることができる分散を示します。
各値は各主成分の分散に等しく、配列の長さは n_components で定義された成分の数に等しくなります。
[13]:
print("寄与率:", pca.explained_variance_ratio_) # 寄与率
print("累積寄与率:", sum(pca.explained_variance_ratio_)) # 累積寄与率
寄与率: [0.72962445 0.22850762]
累積寄与率: 0.9581320720000163
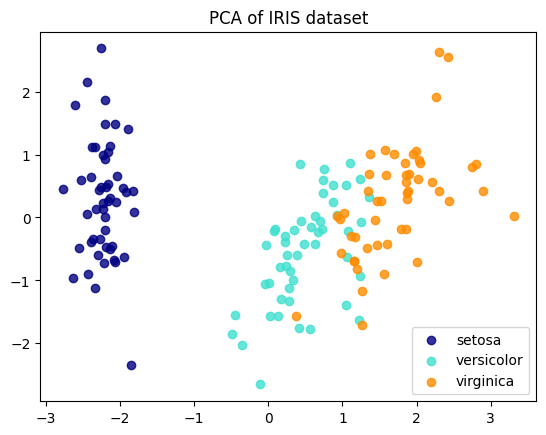
PCA後のデータを可視化
[14]:
colors = ['navy', 'turquoise', 'darkorange']
target_names = ['setosa','versicolor','virginica']
for color, i, target_name in zip(colors, [0, 1, 2], target_names):
plt.scatter(X_pca[y == i, 0], X_pca[y == i, 1], color=color, alpha=.8, label=target_name)
plt.legend(loc='best', shadow=False, scatterpoints=1)
plt.title('PCA of IRIS dataset')
plt.show()